如果您想了解提高熊猫的相关知识,那么本文是一篇不可错过的文章,我们将对PyTables?HDF5表的写入性能进行全面详尽的解释,并且为您提供关于99乘法表的正反写(python的写法)、HDF5文件及
如果您想了解提高熊猫的相关知识,那么本文是一篇不可错过的文章,我们将对PyTables?HDF5表的写入性能进行全面详尽的解释,并且为您提供关于99乘法表的正反写 (python的写法)、HDF5 文件及Python模块之h5py、hdfs深入:06、hdfs的写入过程、IBInspectable / IBDesignable (只能用于Swift?)的有价值的信息。
本文目录一览:- 提高熊猫(PyTables?)HDF5表的写入性能
- 99乘法表的正反写 (python的写法)
- HDF5 文件及Python模块之h5py
- hdfs深入:06、hdfs的写入过程
- IBInspectable / IBDesignable (只能用于Swift?)

提高熊猫(PyTables?)HDF5表的写入性能
我已经使用熊猫进行研究了大约两个月,取得了很大的效果。通过大量的中型跟踪事件数据集,pandas +
PyTables(HDF5接口)在允许我使用我所熟悉和喜爱的所有Python工具处理异构数据方面发挥了巨大作用。
一般来说,我在PyTables中使用“固定”(以前称为“存储”)格式,因为我的工作流程是一次写入,多次读取,并且我的许多数据集都具有一定的大小,因此我可以将其中的50-100个加载到内存中。时间没有严重的不利。(注意:我大部分工作都是在128GB以上系统内存的Opteron服务器级计算机上进行的。)
但是,对于大型数据集(500MB或更大),我希望能够使用PyTables“表”格式的更具扩展性的随机访问和查询功能,以便我可以在内存外执行查询,然后将小得多的结果集加载到内存中进行处理。但是,这里最大的障碍是写入性能。是的,正如我所说,我的工作流程是一次写入,多次读取,但是相对时间仍然不可接受。
举例来说,我最近在48台核心计算机上运行了一个大型的Cholesky因式分解,花费了3分8秒(188秒)。这样就生成了一个约2.2 GB的跟踪文件-
该跟踪是与程序并行生成的,因此没有额外的“跟踪创建时间”。
我的二进制跟踪文件最初转换为pandas /
PyTables格式需要花费大量的时间,但是很大程度上是因为二进制格式故意乱序,以降低跟踪生成器本身的性能影响。从存储格式转换为表格格式时,这也与性能损失无关。
我的测试最初是使用pandas 0.12,numpy 1.7.1,PyTables 2.4.0和numexpr
0.20.1运行的。我的48核计算机每核运行2.8 GHz,我正在写一个ext3文件系统,该文件系统可能(但不一定)在SSD上。
我可以在7.1秒内将整个数据集写入存储格式的HDF5文件(结果文件大小:3.3GB)。写入表格式的相同数据集(结果文件大小也为3.3GB),需要178.7秒的时间来写入。
代码如下:
with Timer() as t: store = pd.HDFStore(''test_storer.h5'', ''w'') store.put(''events'', events_dataset, table=False, append=False)print(''Fixed format write took '' + str(t.interval))with Timer() as t: store = pd.HDFStore(''test_table.h5'', ''w'') store.put(''events'', events_dataset, table=True, append=False)print(''Table format write took '' + str(t.interval))输出是简单的
Fixed format write took 7.1Table format write took 178.7我的数据集有28,880,943行,而列是基本数据类型:
node_id int64thread_id int64handle_id int64type int64begin int64end int64duration int64flags int64unique_id int64id int64DSTL_LS_FULL float64L2_DMISS float64L3_MISS float64kernel_type float64dtype: object…所以我认为写入速度不应该有任何特定于数据的问题。
我还尝试添加BLOSC压缩,以排除可能影响一种情况或另一种情况的任何奇怪的I / O问题,但是压缩似乎同样降低了这两种情况的性能。
现在,我意识到pandas文档说Storer格式提供了更快的写入速度和更快的读取速度。(我确实体验到了更快的读取速度,因为读取Storer格式似乎需要大约2.5秒,而读取Table格式大约需要10秒。)但是,将Table格式写入的时间实际上需要25倍,这似乎确实过高了。只要存储格式写入。
涉及PyTables或Pandas的任何人都可以解释在架构上(或其他方面)写可查询格式(显然只需要很少的额外数据)需要花费一个数量级以上的原因吗?将来有没有改善的希望?我很乐意为一个或另一个项目做出贡献,因为我的领域是高性能计算,并且我看到该领域中两个项目都有着重要的用例…。但是,这对于澄清一些内容将很有帮助。那些首先涉及的问题,和/或一些了解如何构建系统的人提出的有关如何加快运行速度的建议。
编辑:
在IPython中使用%prun运行先前的测试,将为Storer / Fixed格式提供以下(为可读性而有所减少的)概要文件输出:
%prun -l 20 profile.events.to_hdf(''test.h5'', ''events'', table=False, append=False)3223 function calls (3222 primitive calls) in 7.385 secondsOrdered by: internal timeList reduced from 208 to 20 due to restriction <20>ncalls tottime percall cumtime percall filename:lineno(function) 6 7.127 1.188 7.128 1.188 {method ''_createArray'' of ''tables.hdf5Extension.Array'' objects} 1 0.242 0.242 0.242 0.242 {method ''_closeFile'' of ''tables.hdf5Extension.File'' objects} 1 0.003 0.003 0.003 0.003 {method ''_g_new'' of ''tables.hdf5Extension.File'' objects} 46 0.001 0.000 0.001 0.000 {method ''reduce'' of ''numpy.ufunc'' objects}以及以下表格格式:
%prun -l 40 profile.events.to_hdf(''test.h5'', ''events'', table=True, append=False, chunksize=1000000) 499082 function calls (499040 primitive calls) in 188.981 seconds Ordered by: internal time List reduced from 526 to 40 due to restriction <40> ncalls tottime percall cumtime percall filename:lineno(function) 29 92.018 3.173 92.018 3.173 {pandas.lib.create_hdf_rows_2d} 640 20.987 0.033 20.987 0.033 {method ''_append'' of ''tables.hdf5Extension.Array'' objects} 29 19.256 0.664 19.256 0.664 {method ''_append_records'' of ''tables.tableExtension.Table'' objects} 406 19.182 0.047 19.182 0.047 {method ''_g_writeSlice'' of ''tables.hdf5Extension.Array'' objects} 14244 10.646 0.001 10.646 0.001 {method ''_g_readSlice'' of ''tables.hdf5Extension.Array'' objects} 472 10.359 0.022 10.359 0.022 {method ''copy'' of ''numpy.ndarray'' objects} 80 3.409 0.043 3.409 0.043 {tables.indexesExtension.keysort} 2 3.023 1.512 3.023 1.512 common.py:134(_isnull_ndarraylike) 41 2.489 0.061 2.533 0.062 {method ''_fillCol'' of ''tables.tableExtension.Row'' objects} 87 2.401 0.028 2.401 0.028 {method ''astype'' of ''numpy.ndarray'' objects} 30 1.880 0.063 1.880 0.063 {method ''_g_flush'' of ''tables.hdf5Extension.Leaf'' objects} 282 0.824 0.003 0.824 0.003 {method ''reduce'' of ''numpy.ufunc'' objects} 41 0.537 0.013 0.668 0.016 index.py:607(final_idx32) 14490 0.385 0.000 0.712 0.000 array.py:342(_interpret_indexing) 39 0.279 0.007 19.635 0.503 index.py:1219(reorder_slice) 2 0.256 0.128 10.063 5.031 index.py:1099(get_neworder) 1 0.090 0.090 119.392 119.392 pytables.py:3016(write_data) 57842 0.087 0.000 0.087 0.000 {numpy.core.multiarray.empty} 28570 0.062 0.000 0.107 0.000 utils.py:42(is_idx) 14164 0.062 0.000 7.181 0.001 array.py:711(_readSlice)编辑2:
再次使用熊猫0.13的预发行副本运行(2013年11月20日在美国东部时间11:00左右拉出),Tables格式的写入时间显着改善,但仍无法“合理地”与Storer
/ Fixed的写入速度进行比较格式。
%prun -l 40 profile.events.to_hdf(''test.h5'', ''events'', table=True, append=False, chunksize=1000000) 499748 function calls (499720 primitive calls) in 117.187 seconds Ordered by: internal time List reduced from 539 to 20 due to restriction <20> ncalls tottime percall cumtime percall filename:lineno(function) 640 22.010 0.034 22.010 0.034 {method ''_append'' of ''tables.hdf5Extension.Array'' objects} 29 20.782 0.717 20.782 0.717 {method ''_append_records'' of ''tables.tableExtension.Table'' objects} 406 19.248 0.047 19.248 0.047 {method ''_g_writeSlice'' of ''tables.hdf5Extension.Array'' objects} 14244 10.685 0.001 10.685 0.001 {method ''_g_readSlice'' of ''tables.hdf5Extension.Array'' objects} 472 10.439 0.022 10.439 0.022 {method ''copy'' of ''numpy.ndarray'' objects} 30 7.356 0.245 7.356 0.245 {method ''_g_flush'' of ''tables.hdf5Extension.Leaf'' objects} 29 7.161 0.247 37.609 1.297 pytables.py:3498(write_data_chunk) 2 3.888 1.944 3.888 1.944 common.py:197(_isnull_ndarraylike) 80 3.581 0.045 3.581 0.045 {tables.indexesExtension.keysort} 41 3.248 0.079 3.294 0.080 {method ''_fillCol'' of ''tables.tableExtension.Row'' objects} 34 2.744 0.081 2.744 0.081 {method ''ravel'' of ''numpy.ndarray'' objects} 115 2.591 0.023 2.591 0.023 {method ''astype'' of ''numpy.ndarray'' objects} 270 0.875 0.003 0.875 0.003 {method ''reduce'' of ''numpy.ufunc'' objects} 41 0.560 0.014 0.732 0.018 index.py:607(final_idx32) 14490 0.387 0.000 0.712 0.000 array.py:342(_interpret_indexing) 39 0.303 0.008 19.617 0.503 index.py:1219(reorder_slice) 2 0.288 0.144 10.299 5.149 index.py:1099(get_neworder) 57871 0.087 0.000 0.087 0.000 {numpy.core.multiarray.empty} 1 0.084 0.084 45.266 45.266 pytables.py:3424(write_data) 1 0.080 0.080 55.542 55.542 pytables.py:3385(write)我在运行这些测试时注意到,在很长一段时间内写入似乎“暂停”(磁盘上的文件没有活跃增长),但在某些时期内CPU使用率也很低。
我开始怀疑某些已知的ext3限制可能与pandas或PyTables交互不良。例如,Ext3和其他基于非扩展的文件系统有时难以迅速取消链接大文件,并且即使在简单的“
rm” 1GB文件的过程中,类似的系统性能(较低的CPU使用率,但等待时间较长)也很明显。
为了澄清,在每个测试案例中,我确保在开始测试之前删除了现有文件(如果有),以免引起ext3文件删除/覆盖损失。
但是,当使用index =
None重新运行此测试时,性能将大大提高(索引时约为50秒,而索引时约为120秒)。因此,似乎该过程继续受CPU限制(我的系统具有运行在2.8GHz上的相对较旧的AMD
Opteron Istanbul
CPU,尽管它也确实有8个插槽,每个插槽中都有6个核心CPU,但除了一个之外,其他所有插槽都当然,在写入过程中请保持闲置状态),或者在已经部分或完全在文件系统上的情况下,PyTables或pandas尝试操纵/读取/分析文件的方式之间存在一些冲突,导致在建立索引时出现病理上不良的I
/ O行为发生。
编辑3:
在将PyTables从2.4升级到3.0.0之后,@ Jeff建议在较小的数据集(磁盘上为1.3 GB)上进行测试,这使我明白了:
In [7]: %timeit f(df)1 loops, best of 3: 3.7 s per loopIn [8]: %timeit f2(df) # where chunksize= 2 000 0001 loops, best of 3: 13.8 s per loopIn [9]: %timeit f3(df) # where chunksize= 2 000 0001 loops, best of 3: 43.4 s per loop实际上,除了打开索引功能(默认设置)外,我的性能在所有情况下似乎都胜过他。但是,索引编制似乎仍然是一个杀手er,如果我解释这些测试的结果top以及ls在运行这些测试时是正确的,那么仍有一段时间不会进行大量处理,也不会进行任何文件写入(例如,CPU)
Python进程的使用率接近0,并且文件大小保持不变)。我只能假设这些是文件读取。我很难理解为什么文件读取会导致速度变慢,因为我可以在3秒内将整个3+
GB的文件从该磁盘可靠地加载到内存中。如果不是文件读取,那么系统在“等待”什么?(没有其他人登录到计算机中,并且没有其他文件系统活动。)
此时,使用相关python模块的升级版本,原始数据集的性能下降到下图。特别令人感兴趣的是系统时间(我认为这至少是执行IO所花费的时间的上限)和墙时间(Wall
time),这似乎可以解释这些无写/无CPU活动的神秘时期。
In [28]: %time f(profile.events)CPU times: user 0 ns, sys: 7.16 s, total: 7.16 sWall time: 7.51 sIn [29]: %time f2(profile.events)CPU times: user 18.7 s, sys: 14 s, total: 32.7 sWall time: 47.2 sIn [31]: %time f3(profile.events)CPU times: user 1min 18s, sys: 14.4 s, total: 1min 32sWall time: 2min 5s不过,对于我的用例而言,索引似乎会导致速度显着下降。也许我应该尝试限制索引的字段,而不是简单地执行默认情况(这很可能是对DataFrame中所有字段的索引)?我不确定这将如何影响查询时间,尤其是在查询是基于非索引字段进行选择的情况下。
根据Jeff的请求,生成文件的ptdump。
ptdump -av test.h5/ (RootGroup) '''' /._v_attrs (AttributeSet), 4 attributes: [CLASS := ''GROUP'', PYTABLES_FORMAT_VERSION := ''2.1'', TITLE := '''', VERSION := ''1.0'']/df (Group) '''' /df._v_attrs (AttributeSet), 14 attributes: [CLASS := ''GROUP'', TITLE := '''', VERSION := ''1.0'', data_columns := [], encoding := None, index_cols := [(0, ''index'')], info := {1: {''type'': ''Index'', ''names'': [None]}, ''index'': {}}, levels := 1, nan_rep := ''nan'', non_index_axes := [(1, [''node_id'', ''thread_id'', ''handle_id'', ''type'', ''begin'', ''end'', ''duration'', ''flags'', ''unique_id'', ''id'', ''DSTL_LS_FULL'', ''L2_DMISS'', ''L3_MISS'', ''kernel_type''])], pandas_type := ''frame_table'', pandas_version := ''0.10.1'', table_type := ''appendable_frame'', values_cols := [''values_block_0'', ''values_block_1'']]/df/table (Table(28880943,)) '''' description := { "index": Int64Col(shape=(), dflt=0, pos=0), "values_block_0": Int64Col(shape=(10,), dflt=0, pos=1), "values_block_1": Float64Col(shape=(4,), dflt=0.0, pos=2)} byteorder := ''little'' chunkshape := (4369,) autoindex := True colindexes := { "index": Index(6, medium, shuffle, zlib(1)).is_csi=False} /df/table._v_attrs (AttributeSet), 15 attributes: [CLASS := ''TABLE'', FIELD_0_FILL := 0, FIELD_0_NAME := ''index'', FIELD_1_FILL := 0, FIELD_1_NAME := ''values_block_0'', FIELD_2_FILL := 0.0, FIELD_2_NAME := ''values_block_1'', NROWS := 28880943, TITLE := '''', VERSION := ''2.7'', index_kind := ''integer'', values_block_0_dtype := ''int64'', values_block_0_kind := [''node_id'', ''thread_id'', ''handle_id'', ''type'', ''begin'', ''end'', ''duration'', ''flags'', ''unique_id'', ''id''], values_block_1_dtype := ''float64'', values_block_1_kind := [''DSTL_LS_FULL'', ''L2_DMISS'', ''L3_MISS'', ''kernel_type'']]另一个%prun与更新的模块和完整的数据集:
%prun -l 25 %time f3(profile.events)CPU times: user 1min 14s, sys: 16.2 s, total: 1min 30sWall time: 1min 48s 542678 function calls (542650 primitive calls) in 108.678 seconds Ordered by: internal time List reduced from 629 to 25 due to restriction <25> ncalls tottime percall cumtime percall filename:lineno(function) 640 23.633 0.037 23.633 0.037 {method ''_append'' of ''tables.hdf5extension.Array'' objects} 15 20.852 1.390 20.852 1.390 {method ''_append_records'' of ''tables.tableextension.Table'' objects} 406 19.584 0.048 19.584 0.048 {method ''_g_write_slice'' of ''tables.hdf5extension.Array'' objects} 14244 10.591 0.001 10.591 0.001 {method ''_g_read_slice'' of ''tables.hdf5extension.Array'' objects} 458 9.693 0.021 9.693 0.021 {method ''copy'' of ''numpy.ndarray'' objects} 15 6.350 0.423 30.989 2.066 pytables.py:3498(write_data_chunk) 80 3.496 0.044 3.496 0.044 {tables.indexesextension.keysort} 41 3.335 0.081 3.376 0.082 {method ''_fill_col'' of ''tables.tableextension.Row'' objects} 20 2.551 0.128 2.551 0.128 {method ''ravel'' of ''numpy.ndarray'' objects} 101 2.449 0.024 2.449 0.024 {method ''astype'' of ''numpy.ndarray'' objects} 16 1.789 0.112 1.789 0.112 {method ''_g_flush'' of ''tables.hdf5extension.Leaf'' objects} 2 1.728 0.864 1.728 0.864 common.py:197(_isnull_ndarraylike) 41 0.586 0.014 0.842 0.021 index.py:637(final_idx32) 14490 0.292 0.000 0.616 0.000 array.py:368(_interpret_indexing) 2 0.283 0.142 10.267 5.134 index.py:1158(get_neworder) 274 0.251 0.001 0.251 0.001 {method ''reduce'' of ''numpy.ufunc'' objects} 39 0.174 0.004 19.373 0.497 index.py:1280(reorder_slice) 57857 0.085 0.000 0.085 0.000 {numpy.core.multiarray.empty} 1 0.083 0.083 35.657 35.657 pytables.py:3424(write_data) 1 0.065 0.065 45.338 45.338 pytables.py:3385(write) 14164 0.065 0.000 7.831 0.001 array.py:615(__getitem__) 28570 0.062 0.000 0.108 0.000 utils.py:47(is_idx) 47 0.055 0.001 0.055 0.001 {numpy.core.multiarray.arange} 28570 0.050 0.000 0.090 0.000 leaf.py:397(_process_range) 87797 0.048 0.000 0.048 0.000 {isinstance}答案1
小编典典那是一个有趣的讨论。我认为Peter可以在固定格式上获得出色的性能,因为该格式可以一次性写入,并且他具有非常好的SSD(可以以超过450 MB /
s的速度写入)。
追加到表是一个更复杂的操作(必须扩大数据集,并且必须检查新记录,以便我们可以确保它们遵循表的架构)。这就是为什么在表中附加行通常较慢的原因(但Jeff的速度约为70
MB / s,这是相当不错的)。Jeff比Peter获得更快的速度可能是由于他拥有更好的处理器。
最后,在PyTables中建立索引是使用单个处理器,是的,这通常是一项昂贵的操作,因此,如果您不打算在磁盘上查询数据,则应该禁用它。

99乘法表的正反写 (python的写法)
# 正写
j = 0
k = 0
while j < 10:
j+=1
while k <10:
k+=1
if j>k:
k = 0
break
else:
print(''{}*{}={}''.format(j,k,j*k))
print()
# 反写
j = 10
k = 10
while 0<j:
j -=1
while 1<k:
k-=1
if k == 0
k = j
break
else:
print(''{}*{}={}''.format(j,k,j*k))
print()/print('''')

HDF5 文件及Python模块之h5py
HDF5文件
什么是HDF5文件呢?先引用一波维基百科的介绍,『层级数据格式(Hierarchical Data Format:HDF)是设计用来存储和组织大量数据的一组文件格式(HDF4,HDF5)。它最初开发于美国国家超级计算应用中心,现在由非营利社团HDF Group支持,其任务是确保HDF5技术的持续开发和存储在HDF中数据的持续可访问性。』。HDF5 拥有一系列的优异特性,使其特别适合进行大量科学数据的存储和操作,如它支持非常多的数据类型,灵活,通用,跨平台,可扩展,高效的 I/O 性能,支持几乎无限量(高达 EB)的单文件存储等
如何在Linux中查看hdf5文件呢?
h5ls info.h5
# key1 Dataset {10000}
# key2 Dataset {10000,5}
# key3 Dataset {20000,30}
h5py模块
我们可以使用Python非常方便的读写hdf5文件,最常用的模块就是h5py。下面说明一下它的安装及使用方法:
安装模块
pip install h5py
pip install numpy
# numpy 通常是作为配合使用
这里引用一个博主对h5py的总结(https://www.jianshu.com/p/de9f33cdfba0):
『一个 HDF5 文件是存储两类对象的容器,这两类对象分别为:
dataset:类似数组的数据集合; gropp;类似目录的容器,其中可以包含一个或多个 dataset 及其它的 group。
一个 HDF5 文件从一个命名为 "/" 的 group 开始,所有的 dataset 和其它 group 都包含在此 group 下,当操作 HDF5 文件时,如果没有显式指定 group 的 dataset 都是默认指 "/" 下的 dataset,另外类似相对文件路径的 group 名字都是相对于 "/" 的。
HDF5 文件的 dataset 和 group 都可以拥有描述性的元数据,称作 attribute。
用 h5py 操作 HDF5 文件,我们可以像使用目录一样使用 group,像使用 numpy 数组一样使用 dataset,像使用字典一样使用属性,非常方便和易用。』
写入hdf5文件
import h5py
import numpy as np
# 如果你要在根group下创建dataset
f = h5py.File(''info.h5'', ''w'')
values1 = np.arange(12).reshape(4, 3)
values2 = np.arange(20).reshape(4, 5)
f.create_dataset(name=''key1'', data=np.array(values1, dtype=''int64''))
f.create_dataset(name=''key2'', data=np.array(values2, dtype=''int64''))
# 如果你要创建一个group(目录)
# 然后指定dataset放置的group
f.create_group(''/dir1'')
f.create_group(''/dir1/dir2'')
data = np.arange(6).reshape(3, 2)
f.create_dataset(''/dir1/dir2'', data=data)
# 最后别忘了关闭文件
f.close()
读取hdf5文件
import h5py
with h5py.File(info.h5, ''r'') as f:
values1 = f[''key1''].value
values2 = f[''key2''].value

hdfs深入:06、hdfs的写入过程
7、HDFS的文件写入过程
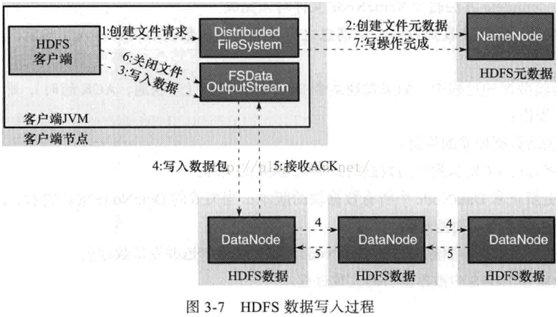
详细步骤解析:
1、 client发起文件上传请求,通过RPC与NameNode建立通讯,NameNode检查目标文件是否已存在,父目录是否存在,返回是否可以上传;
2、 client请求第一个block该传输到哪些DataNode服务器上;
3、 NameNode根据配置文件中指定的备份数量及机架感知原理进行文件分配,返回可用的DataNode的地址如:A,B,C;
注:Hadoop在设计时考虑到数据的安全与高效,数据文件默认在HDFS上存放三份,存储策略为本地一份,同机架内其它某一节点上一份,不同机架的某一节点上一份。
4、 client请求3台DataNode中的一台A上传数据(本质上是一个RPC调用,建立pipeline),A收到请求会继续调用B,然后B调用C,将整个pipeline建立完成,后逐级返回client;
5、 client开始往A上传第一个block(先从磁盘读取数据放到一个本地内存缓存),以packet为单位(默认64K),A收到一个packet就会传给B,B传给C;A每传一个packet会放入一个应答队列等待应答。
6、 数据被分割成一个个packet数据包在pipeline上依次传输,在pipeline反方向上,逐个发送ack(命令正确应答),最终由pipeline中第一个DataNode节点A将pipelineack发送给client;
7、 当一个block传输完成之后,client再次请求NameNode上传第二个block到服务器。
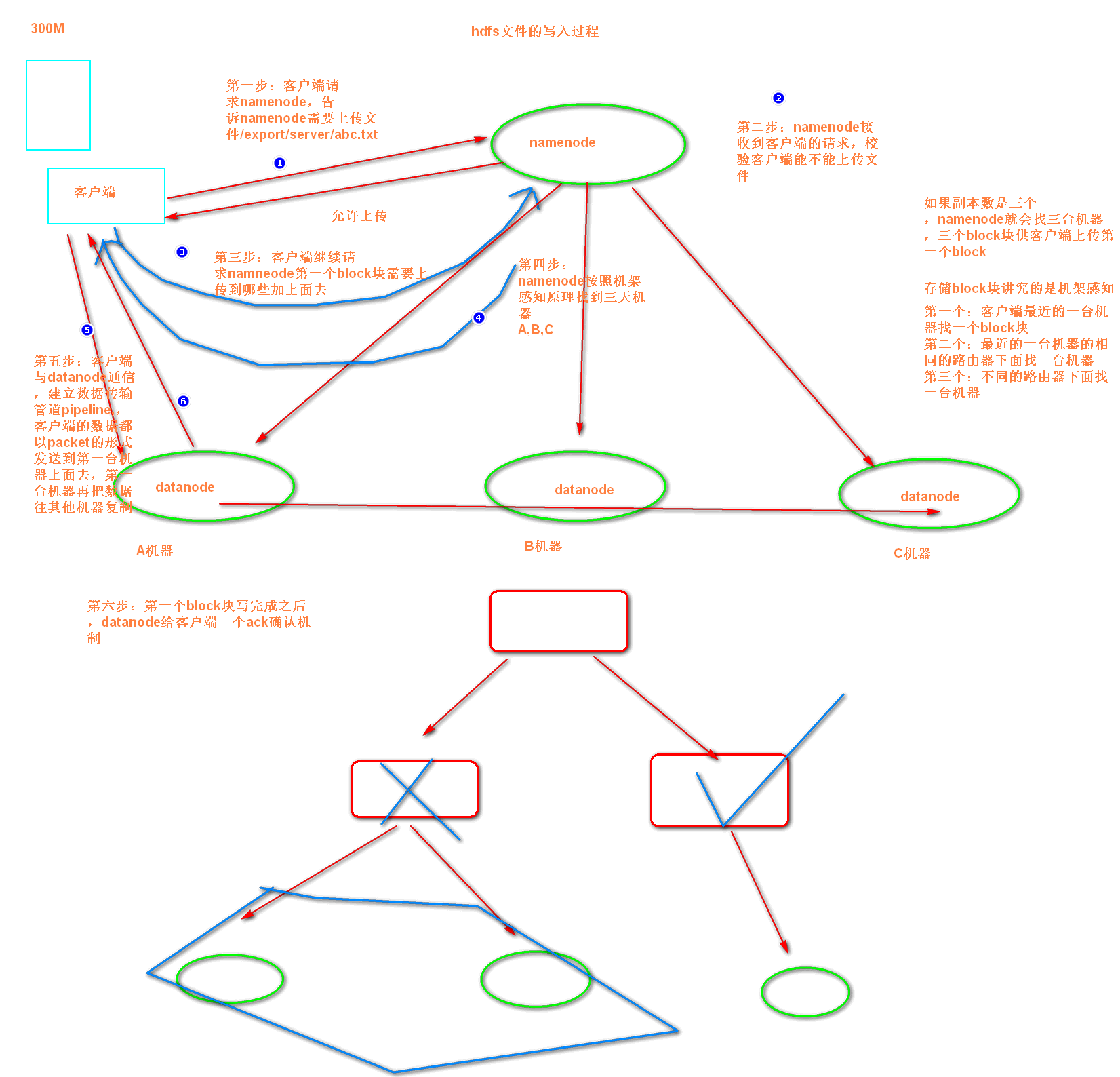
hdfs的文件写入过程(课上老师的总结):
第一步:客户端发出请求,请求namneode需要上传数据
第二步:namenode检测客户端是或否有权限上传
第三步:客户端请求namenode第一个block块上传到哪里去
第四步:namenode找三个block块返回给客户端
第五步:客户端找datanode建立pipeline管道,主备上传数据,数据都是以packet包的形式通过管道上传到datanode上面去
第六步:datanode保存好了之后,给客户端一个ack确认机制,客户端准备上传下一个block块,直到所有的block块上传完成,关闭文件流

IBInspectable / IBDesignable (只能用于Swift?)
IBInspectable
IBInspectable 属性提供了访问旧功能的新方式:用户自定义的运行时属性。从目前的身份检查器(identity inspector)中访问,这些属性在 Interface Builder 被整合到 Xcode 之前就可用了。他们提供了一个强有力的机制来配置一个 NIB,XIB,或者 storyboard 实例中的任何键值编码(key-value coded)属性:
虽然功能强大,运行时属性可能会使工作很繁琐。一个属性的关键字路径,类型和属性值需要在每个实例设置,没有任何自动完成或输入提示,这就需要前往文档或自定义子类的源代码仔细检查设置。 IBInspectable 属性彻底的解决了这个问题:在 Xcode 6,你现在可以指定任何属性作为可检查项并为你的自定义类建立了一个用户界面。
例如,在一个 UIView 子类里,这些属性用它们的值来更新背景层:
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
|
@IBInspectable
var
cornerRadius:CGFloat=0{
didSet{
layer.cornerRadius=cornerRadius
layer.masksToBounds=cornerRadius>0
}
}
borderWidth:CGFloat=0{
didSet{
layer.borderWidth=borderWidth
}
}
borderColor:UIColor?{
didSet{
layer.borderColor=borderColor?.CGColor
}
}
|
标有 @IBInspectable(或是 Objective-C 中的 IBInspectable),他们就可以很容易在 Interface Builder 的观察面板(inspector panel)里编辑。需要注意的是 Xcode 在这里做了更多的事,属性名称是从 camel- 转换为 title- 模式 并且相关的名称组合在一起:
因为可检查属性仅仅是用户定义的运行时属性顶部的接口,所以支持相同的类型列表:布尔,字符串和数字(即,NSNumber 或任何数值类型),以及 CGPoint、CGSize、CGRect、UIColor 和 NSRange,额外增加了 UIImage。
那些已经熟悉运行时属性的人将注意到在上面的例子中有一些问题。UIColor 是里面唯一支持色彩的类型,而不是原生支持视图 CALayer 的 CGColor。borderColor 会计算 UIColor 属性(通过运行时属性设置)并映射到该层需要的 CGColor。
让现有的类型可观察
内置的 Cocoa 类型如果在 Interface Builder 中的属性检查器中没有列出也可以通过扩展来使属性可视。如果你喜欢圆角,你一定会喜欢这个 UIView 扩展:
extensionUIView{
cornerRadius:CGFloat{
get{
return
layer.cornerRadius
}
set{
layer.cornerRadius=newValue
layer.masksToBounds=newValue>0
}
变!你创建的任何 UIView 都将有一个可配置的边界半径。
|
今天关于提高熊猫和PyTables?HDF5表的写入性能的讲解已经结束,谢谢您的阅读,如果想了解更多关于99乘法表的正反写 (python的写法)、HDF5 文件及Python模块之h5py、hdfs深入:06、hdfs的写入过程、IBInspectable / IBDesignable (只能用于Swift?)的相关知识,请在本站搜索。
本文标签:



![[转帖]Ubuntu 安装 Wine方法(ubuntu如何安装wine)](https://www.gvkun.com/zb_users/cache/thumbs/4c83df0e2303284d68480d1b1378581d-180-120-1.jpg)

